<研究の背景と経緯>
ロバストネス(頑健性)は、さまざまな摂動(外乱・内乱)に抗って機能を維持しようとするシステムの特性で、生命現象に普遍的に見られる特徴です。これは、淘汰圧をうけながら進化してきた複雑なシステムである生命システムの基本特性と考えられてきました。生命のロバストネスは、生命をシステムレベルで扱ったときのみそれを実現するための原理の議論が可能になるため、近年急速に発展しているシステムレベルでの生命研究(システムバイオロジー)によりようやくその原理が理解されるようになってきました。生物のロバストネスを知ることで生命システムの基本原理の解明、コンピュータ細胞モデルの開発、疾患に対する新たな治療手段の提供が可能になることが提唱されています。
これまでの生命のロバストネスについての研究はそのほとんどが理論的な研究にとどまっていました。これは、実際の細胞のロバストネスを検定する有効な実験技術がなかったことがその大きな原因のひとつです。ロバストネスに関する理論的な研究を実践的・実用的な研究に発展させるためには、このような実験技術は必ず必要となるものでした。そこで本研究では実際の細胞システムのもつロバストネスを検定する技術を開発し、そこで得られる全く新しいタイプのデータセット(ロバストネス・プロファイル)を用いて生命のシステムレベルの理解を深めることを目的としました。
<研究の内容>
- 本研究では、遺伝子過剰発現という摂動に対する細胞のロバストネスを検定する新しい実験法として、gTOW法を考案・開発しました。この手法では、遺伝学的な選択によって増殖を維持するための遺伝子コピー数の上限を決定することが可能です。
- この手法を出芽酵母の細胞周期制御にかかわる30の遺伝子(CDC遺伝子)に適応し、それらの遺伝子コピー数の上限値を測定しました。これらは2コピーから100コピーでした。これは遺伝子のコピーの上限値を測定した初めての例です。
- 測定データをシステム全体として提示したものをロバストネス・プロファイルと名づけました。これを細胞周期の分子相互作用ネットワーク上にあらわしたものが図1です。この結果、出芽酵母の細胞周期ではB型サイクリン依存性キナーゼの制御を行う遺伝子群を含むサブシステムの脆弱性が特に高い事が明らかになりました(図1)。このロバストネス・プロファイルにより細胞システムのロバストな部分と脆弱な部分が浮き彫りにされました。
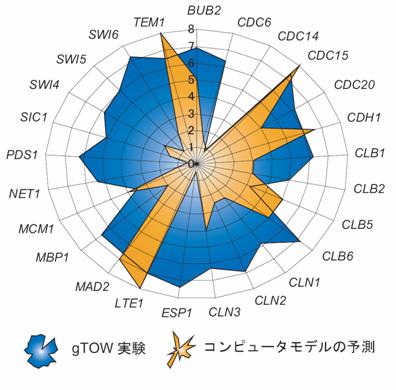
- ロバストネス・プロファイルを用いて細胞周期のコンピュータモデルの予測値とgTOW法で得られたCDC遺伝子のコピー数の上限値との比較を行い、コンピュータモデルの確からしさを一度に検証することに成功しました(図2)。
<今後の展開>
本研究ではモデル生物である出芽酵母を使って、細胞のロバストネスを実際に検定するためにはどのような実験手法が有効なのか、またロバストネスを検定するが生命システムの理解に如何に貢献できるのかを示すことができました。今後は本研究で作られたロバストネス研究の枠組みを利用して、細胞システムのロバストネスを検定することを目的とした様々な研究手法が生まれ、細胞システムの理解が深まると思われます。
また今後の応用面でのひとつの大きな展開として、健常な細胞と腫瘍細胞のロバストネス・プロファイルを取得し、腫瘍細胞のみが持つ脆弱点をあぶりだし、そこを新しい薬剤の標的とするといった、ロバストネスを指標にした新しい治療手段の構築が期待されます。
【用語解説】
- 細胞周期
- 細胞の増殖と分裂をつかさどるシステム。細胞は細胞周期の1サイクルでG1期、S期、G2期、M期という4つの期を通して、染色体を倍加させそれを正確に分配させて2つの細胞を作ります。出芽酵母では細胞周期の分子メカニズムがもっとも詳細に研究されています。図1に示したものはこれらの知識を統合して作られた分子相互作用ネットワークです。細胞周期の異常は細胞の癌化につながると考えられています。
- 遺伝子過剰発現
- 遺伝子の発現量(通常は遺伝子から読み取られて作られるmRNAや蛋白質の量を指す)は、それぞれの遺伝子によって決まっています。それを人工的な遺伝子操作によって通常の発現量よりも過剰に発現させることを過剰発現と呼びます。本研究では、細胞内の遺伝子の数(遺伝子コピー数)を増やすことによって、遺伝子の過剰発現を引き起こしています。
- ロバストネス・プロファイル
- ある生命システムのロバストネスの特性を提示したもの。本研究では、出芽酵母の細胞周期システムの、遺伝子の過剰発現に対するロバストネスの特性を提示しました(図1)。ロバストネスプロファイルは、様々な生物種、細胞種(臓器の違いや、野生型、変異型細胞などの違い)の持つシステムの特性によって異なっていると考えられます。
- Genetic Tug-of-War(gTOW : 遺伝子綱引き)法
- 本研究で開発された、増殖を阻害するための遺伝子コピー数の上限を網羅的に測定することを可能にする新しい実験手法です。より専門的な解説は図3をご覧ください。
- モデル生物
- 生命現象の基本原理を探るために用いられるより単純な生物。一般的に高等生物よりも優れた実験系を提供します。本研究で用いた出芽酵母は単細胞の真菌で、真核生物としてはもっとも単純ですが、人にまで保存された様々な細胞の機能を備えています。細胞周期もそのような例の一つです。
- 頑健性と脆弱性
- 頑健性(ロバストネス)とは、様々な摂動に抗って機能を維持することのできるシステムの特性を表しています。頑健性(ロバストネス)と脆弱性は相反する関係にあります。すなわち、頑健性が著しく低い場合、脆弱である(脆弱性が高い)と言います。
- コンピュータ細胞モデル
- 微分方程式などを用いて細胞システムの振る舞いをコンピュータ上に再構築したもの。細胞シミュレータもほぼ同じ意味で用いられます。細胞システムの振る舞いを高い精度で予測できるコンピュータモデルが完成すれば、薬剤の効果などをあらかじめシミュレーションによって予測することができると期待されています。
- 遺伝子コピー数
- 細胞内の遺伝子の数。通常、1倍体ゲノムあたり1個です。
- 分子相互作用ネットワーク
- 生命システムを作る分子(遺伝子、蛋白質など)が複雑に相互作用して作るネットワーク。ロバストネスなど細胞のシステムとしての特性はこの相互作用の結果として現れます。
- B型サイクリン依存性キナーゼ
- 細胞周期で中心的な役割を果たしている蛋白質リン酸化酵素(サイクリン依存性キナーゼ)の中で特にB型サイクリンと結合しているもの。S期やM期で重要な働きをすると考えられています。
【研究領域等】
JST戦略的創造研究推進事業 発展研究(SORST)
研究課題名:北野共生システムプロジェクト
総括責任者:北野宏明(ソニーコンピュータサイエンス研究所 取締役副所長)
研究実施場所:慶応義塾大学 医学部
研究実施期間: 平成 16年9月〜
【参考図】
- 図1 出芽酵母細胞周期システムの、遺伝子の過剰発現に対するロバストネス・プロファイル
- 図2 コンピュータモデルと実際の細胞の比較
- 図 3 Genetic Tug-Of-War (gTOW) 法
図1 出芽酵母細胞周期システムの、遺伝子の過剰発現に対するロバストネス・プロファイル 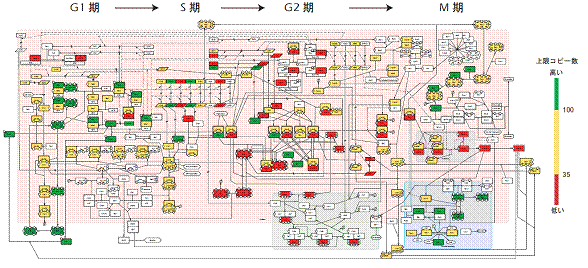
遺伝子コピー数の上限を出芽酵母の分子相互作用マップ上に可視化することで、特に B型サイクリン依存性キナーゼの制御に関わる遺伝子群の上限が低く、この遺伝子群を含むサブシステムの脆弱性が高いことがわかりました。
gTOW法によって得られた遺伝子コピー数の上限(青色)と細胞周期コンピュータモデル(Chen2004)の予測(オレンジ色)との比較をあわらしています。コンピュータモデルは実際の細胞に比べほとんどの遺伝子の上限が低くロバストネスが低いことがわかります。このようにgTOW法を用いてコンピュータモデルの検証を行うことができます。大きく矛盾した部分については未知の因子や制御機構の存在が予想されます。
図 3 Genetic Tug-Of-War (gTOW) 法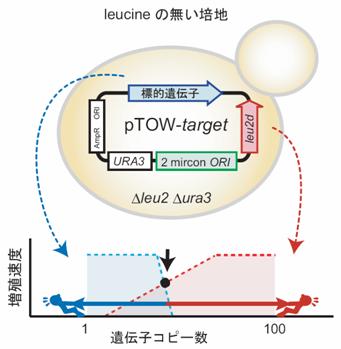
図は、 gTOW法の原理を概念的に表したものです。gTOW法では、 leu2d (非常に活性の低いロイシン合成酵素遺伝子)をもった多コピーのプラスミド上に、標的の遺伝子をクローニングします(左上)。このプラスミドを leu2 遺伝子が破壊された酵母株に導入し、ロイシンのない培地で増殖させると、より高いプラスミドコピー数を持つ細胞がより高い増殖速度を持つために濃縮され(左下、赤い点線のグラフ)、最終的には細胞あたり100コピー以上のプラスミドコピー数をもつ細胞が濃縮されます。すなわち、 leu2d はプラスミドコピー数を100コピー以上に上げようとする遺伝的選択圧として働きます(左下、赤い矢印)。一方、標的の遺伝子が上限をもっている場合、上限より高いプラスミドコピー数を持った細胞の増殖は阻害されます(左下、青い点線のグラフ)。したがって、標的遺伝子はプラスミドコピー数を下げる遺伝的選択圧として働きます(左下、青い矢印)。実際のプラスミドのコピー数は、 leu2d と標的遺伝子の選択圧の引っ張り合いがつりあった数となるわけです。 leu2d の選択圧はどの標的遺伝子を用いた実験でも一定であることから、 このコピー数は標的遺伝子の上限に相関した数となることが予想されます。
実際の細胞内でのプラスミドのコピー数はリアルタイム PCR法によってハプロイドゲノムあたりのコピー数として定量されます。この手法ではネイティブなプロモータをもった遺伝子を基本単位としており、細胞増殖阻害の限界値が「遺伝子コピー数(すなわちネイティブな状態の倍数)」という相対的な数値として得られるため、遺伝子間の比較が可能になります。これは、これまでのプロモーター置換による過剰発現系では得られなかった情報です。